R丨数据分析进阶版
小可爱们好,距离介绍单层模型的R丨数据分析基础版已经过去一年啦。 基础篇主要聚焦于单层模型的检验,介绍了导入原始数据、信效度、描述性统计、相关分析、单层模型的假设检验等等。
这一次我们来介绍跨层模型的数据分析,主要包括ICC、Rwj的计算,跨层模型的假设检验,Monte Carlo估计。
计算Rwj与ICC
Rwj
library(multilevel)
mean(rwg.j(data[,c("X1","X2","X3")], data$GID, 4)$rwg)
median(rwg.j(data[,c("X1","X2","X3")], data$GID, 4)$rwg)
所用R包:multilevel。
核心函数:rwg.j(x, grpid, ranvar)
- x指代题项,如data[,c(“X1”,“X2”,“X3”)],意为后续计算data数据中的X1-X3题项。
- grpid指代分组ID,如data$GID。
- ranvar意为与实际组内方差相比的随机方差,这取决于量纲 ,公式为,所以5点量表填2,7点量表填4。
上述代码第二、三行,分别意味着在【data】中,根据分组ID【GID】,计算7点量表【X1至X3】的均值Rwg或中值Rwg。
Rwj结果
ICC
library(multilevel)
ICC1(aov(X~as.factor(GID), data))
ICC2(aov(X~as.factor(GID), data))
所用R包:multilevel。
核心函数:ICC1(object)、ICC2(object)
上述代码第二、三行,分别意味着在【data】中,根据分组ID【GID】,计算变量【X】的ICC1和ICC2。

跨层模型的假设检验
R进行跨层模型的检验也有多种方式,这里仅介绍萜妹目前最常用的方式:利用MplusAutomation包,在R中进行Mplus分析。MplusAutomation的详细介绍可见:在R中调用Mplus。
我们以最简单的只是数据涉及嵌套,但所有变量均在Level 1的中介模型为例。
library(MplusAutomation)
首先需要加载后续用到的R包。
pathmodel1 <- mplusObject(
TITLE = "Title;",
VARIABLE = "
USEVARIABLES = GID X M Y;
CLUSTER = GID;",
DEFINE= "CENTER X(GroupMEAN);",
ANALYSIS = "TYPE=Complex;",
MODEL = "
M ON X ;
Y on M X;",
OUTPUT = "CINTERVAL standardized Tech1 Tech3;",
rdata = data)
第二步,利用mplusObject函数,在R中创建Mplus模型的输入文件和数据文件。该对象保存了Mplus输入文件的所有部分,以及一些额外的R部分。具体的语句和Mplus里大同小异,额外需要注意的几个点:
- DATA = NULL, !不需要定义,R会自动生成
- VARIABLE = NULL,!不需要定义全部的变量,R自动生成
- rdata = NULL,!定义用于模型的R数据集
fit1 <- mplusModeler(
pathmodel1,
modelout = "Indirect effect.inp",
run = 1L,
writeData = "always",
hashfilename = FALSE)
第三步,利用mplusModeler函数,生成input、data文件,并运行后得到output文件。
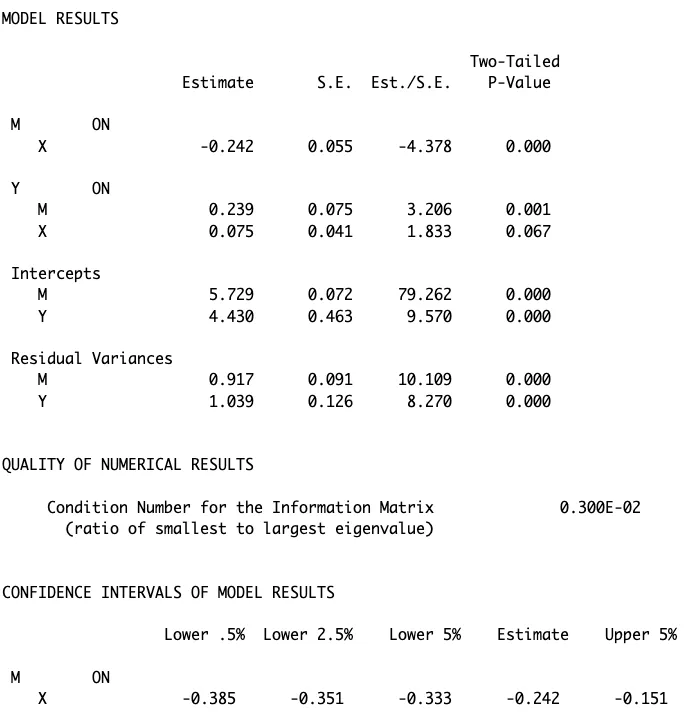
运行后即会生成新的文件,可点击查看结果。


 o
o
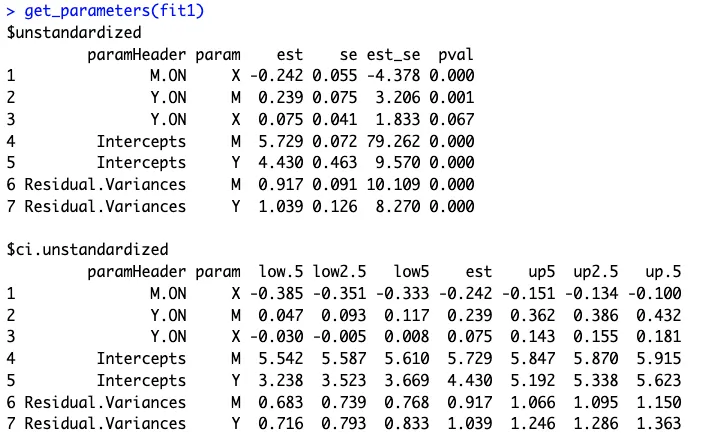
当然,我们也可以利用语句在R中调用结果。
get_parameters(fit1)
Monte Carlo估计
接下来要进行Monte Carlo估计,计算效应的置信区间。
估计所需的数据可以在Mplus的Tech1和Tech3中找到,原理见:如何获得Monte Carlo Bootstrap所需数据。
我们也可以用R呈现Tech1和Tech3结果:
get_tech1(fit1)
get_tech3(fit1)

tech1部分结果

tech3结果
找到相应数据,修改如下语法即可。相关原理与语句意义见如何用R进行跨层效应的Monte Carlo估计。
### 第一部分:数值输入
a=0.1
b=0.2
vara=0.03
varb=0.04
covab=0.005
### 第二部分:进行抽样
rep=20000
pest=c(a,b)
acov <- matrix(c(
vara, covab,
covab, varb
),2,2)
require(MASS)
mcmc <- mvrnorm(rep,pest,acov,empirical=FALSE)
### 第三部分:计算每个抽样的效应值
ab <- mcmc[,1]*mcmc[,2]
### 第四部分:生成置信区间
conf=95
low=(1-conf/100)/2
upp=((1-conf/100)/2)+(conf/100)
LL=quantile(ab,low)
UL=quantile(ab,upp)
LL4=format(LL,digits=4)
UL4=format(UL,digits=4)
### 第五部分:结果可视化
hist(ab,breaks='FD',col='skyblue',xlab=paste(conf,'% Confidence Interval ','LL',LL4,' UL',UL4),
main='Distribution of Indirect Effect')
这篇推送就到这里啦,也算是对之前相关推送的归档,希望能对大家有所帮助。
另外,最近又开始有大活了,只能说我尽力平衡,争取不鸽!
